Archivage de données avec des molécules
Lire plus efficacement les polymères synthétiques comme mémoire de données
De plus en plus de données doivent être stockées, souvent à long terme. Les polymères synthétiques constituent une alternative aux supports de stockage traditionnels, car ils maintiennent les informations stockées avec un besoin d'espace et d'énergie nettement moindre. La lecture des données par spectrométrie de masse limite toutefois la longueur et donc la capacité de stockage des chaînes de polymères individuelles. Dans la revue Angewandte Chemie, une équipe de recherche présente une nouvelle approche qui surmonte cette limitation et permet un accès direct aux bits d'intérêt sans lecture de la chaîne entière.
Wiley-VCH
Des données sont générées quotidiennement, que ce soit dans le cadre de transactions commerciales, de la surveillance des processus, de l'assurance qualité ou de la traçabilité des lots de production. Leur archivage pendant des décennies nécessite beaucoup de place, mais aussi de l'énergie. Les macromolécules à séquence définie, comme l'ADN et les polymères synthétiques, constituent une alternative intéressante, notamment pour l'archivage à long terme de grandes quantités de données auxquelles il n'est que rarement nécessaire d'accéder.
Par rapport à l'ADN, les polymères synthétiques présentent des avantages : une synthèse simple, une densité de stockage plus élevée et une stabilité dans des conditions difficiles. L'inconvénient : l'information codée dans les polymères est lue par spectrométrie de masse (MS) ou par séquençage de masse en tandem (MS2). Pour cela, les molécules ne doivent pas être trop grandes, ce qui limite fortement la capacité de stockage par chaîne. De plus, la chaîne complète est lue bloc par bloc, il n'est pas possible d'accéder directement aux bits intéressants - comme si, au lieu de consulter la page pertinente d'un livre, il fallait le lire en entier. En revanche, les longues chaînes d'ADN peuvent être décomposées en fragments de longueur aléatoire, séquencées individuellement et reconstruites par calcul pour former la séquence globale.
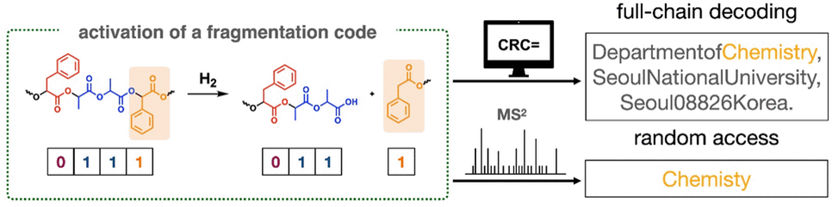
Kyoung Taek Kim et son équipe du département de chimie de l'Université nationale de Séoul (Rep. Corée) ont développé une nouvelle approche permettant de lire efficacement de très longues chaînes de polymères synthétiques dont le poids moléculaire dépasse largement la limite analytique de la MS ou MS2. À titre d'exemple, ils ont codé leur adresse universitaire dans un code ASCII et l'ont traduit - avec un code de détection d'erreur (CRC, méthode courante de vérification de l'intégrité des données) - en un code binaire, c'est-à-dire une séquence de 1 et de 0. Ils ont stocké les informations de 512 bits ainsi générées dans une chaîne de polymère composée de deux monomères différents : L'acide lactique code 1 et l'acide phényl-lactique 0. Ils ont également inséré des codes de fragmentation contenant de l'acide mandélique à des endroits irréguliers. Lors de l'activation chimique, les chaînes y sont scindées, dans l'exemple en 18 fragments de tailles différentes, qui peuvent être décodés individuellement par séquençage MS2.
Un logiciel spécialement développé identifie d'abord les fragments à partir de leur masse ainsi que de leurs groupes terminaux à partir des spectres MS. Pendant la MS2, les ions moléculaires déjà mesurés continuent à se "casser" et les fragments sont à nouveau analysés. Les différences de masse permettent de séquencer les fragments. À l'aide des codes de détection d'erreurs CRC, le logiciel reconstruit la séquence de la chaîne complète. La limitation de la longueur des chaînes de polymères est ainsi surmontée.
En outre, l'équipe a réussi à lire les bits d'intérêt sans séquencer la chaîne polymère complète (accès aléatoire), par exemple le mot "Chemistry" à partir du code pour l'adresse. En tenant compte du fait que toutes les parties de l'adresse sont séparées par des virgules et disposées dans un certain ordre (département, institution, ville, code postal, pays), il a été possible de délimiter l'endroit où l'information recherchée est stockée dans la chaîne et de ne séquencer que les fragments pertinents.
Note: Cet article a été traduit à l'aide d'un système informatique sans intervention humaine. LUMITOS propose ces traductions automatiques pour présenter un plus large éventail d'actualités. Comme cet article a été traduit avec traduction automatique, il est possible qu'il contienne des erreurs de vocabulaire, de syntaxe ou de grammaire. L'article original dans Allemand peut être trouvé ici.