L'imagerie des plus petits atomes permet de comprendre la biochimie inhabituelle d'une enzyme
Cela pourrait contribuer à la conception d'enzymes pour l'industrie chimique
Annonces
Lorsque vos blessures guérissent et que votre foie détoxifie un poison tel que l'histamine que vous avez ingéré, vous pouvez remercier la classe d'enzymes connue sous le nom de cuivre amine oxydase pour son aide. L'identification des positions exactes des plus petits atomes d'hydrogène dans ces enzymes est un défi pour les technologies couramment utilisées, mais elle est essentielle pour concevoir des enzymes améliorées qui présentent une réactivité biochimique inhabituelle mais utile.
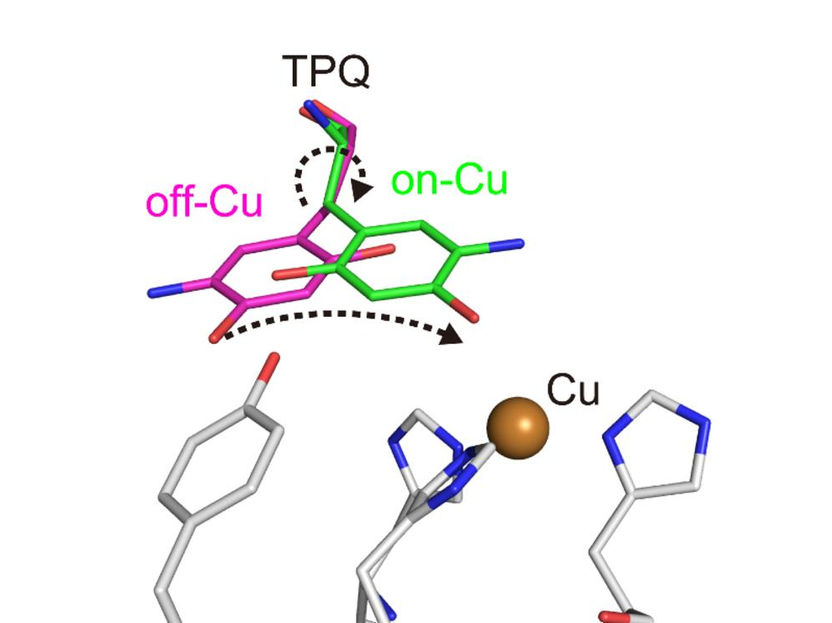
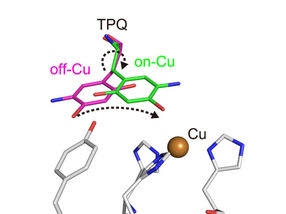
Changement de conformation du cofacteur TPQ au cours de la catalyse. TPQ subit un changement de conformation de off-Cu à on-Cu de manière à générer un intermédiaire radical semiquinone.
Takeshi Murakawa, Toshihide Okajima
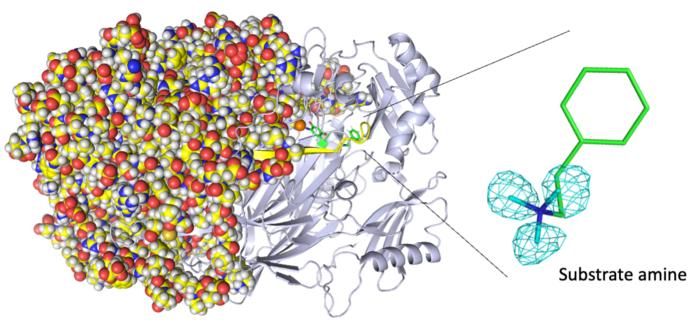
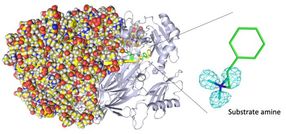
Structure cristalline complète de l'amine oxydase de cuivre. (Gauche) La cristallographie neutronique a permis de déterminer la structure complète de la protéine, y compris les atomes d'hydrogène. (Droite) Les positions précises des atomes d'hydrogène indiquent que la molécule détectée n'est pas un aldéhyde produit mais une amine substrat. Les cartes des atomes d'hydrogène (plus précisément du deutérium) sont présentées à l'aide d'une maille cyan.
Takeshi Murakawa, Toshihide Okajima
Dans une étude récemment publiée dans ACS Catalysis, une équipe dirigée par des chercheurs de l'Université médicale et pharmaceutique d'Osaka et de l'Université d'Osaka a utilisé la cristallographie neutronique pour obtenir une image de la structure atome par atome d'une enzyme cuivre amine oxydase. Cette étude fournit des informations structurelles sans précédent sur la biochimie de l'enzyme.
Certaines enzymes cuivre amine oxydase présentent une biochimie inhabituelle, telle que le tunnel quantique, qui permet des taux de réaction inexplicablement rapides. Bien qu'il soit souvent difficile de déterminer la position exacte de chaque atome d'hydrogène dans l'enzyme, cette connaissance est importante pour concevoir des enzymes artificielles correspondantes. Les chercheurs obtiennent généralement la structure atome par atome des enzymes par cristallographie aux rayons X. Cependant, cette technique ne permet d'obtenir que des informations structurelles. Cependant, cette technique obtient des informations structurelles par diffraction des électrons dans l'enzyme. Elle est donc insuffisante pour imager les atomes d'hydrogène, qui ne contiennent généralement qu'un seul électron. La cristallographie neutronique, qui analyse la diffraction des noyaux atomiques de l'enzyme (tous les atomes ont un noyau atomique), est une technique d'imagerie alternative que les chercheurs ont choisie pour leurs travaux.
"La cristallographie aux rayons X ne permet pas d'expliquer complètement certaines questions relatives à la dépendance au pH, au changement de conformation et à la stabilisation des intermédiaires radicaux de notre enzyme", explique Takeshi Murakawa, auteur principal de l'étude. "La cristallographie neutronique est bien adaptée pour répondre à ces questions".
Les chercheurs ont obtenu de nombreuses informations. Par exemple, ils ont imagé l'état de protonation/déprotonation (lié au pH) des sites de l'enzyme qui sont importants pour stabiliser les espèces radicalaires (c'est-à-dire des atomes particulièrement réactifs qui contiennent un électron non apparié). Ils ont également caractérisé les mouvements du cofacteur topaquinone de l'enzyme - glissement, inclinaison vers le haut et demi-rotation - qui facilitent le transfert d'un seul électron au sein de l'enzyme.
"Nous révélons la liaison d'une deuxième molécule de substrat aminé de haute affinité au cours de la réaction enzymatique, un événement jusqu'alors inconnu dans le site actif de l'enzyme", explique Toshihide Okajima, auteur principal. "La cristallographie aux rayons X ne permet pas d'obtenir de telles informations.
Ce travail a fourni des détails structuraux jusqu'alors inconnus dans une enzyme cuivre amine oxydase qui a de nombreuses fonctions dans le métabolisme biochimique. La révélation de la position exacte des atomes d'hydrogène dans l'enzyme permet d'expliquer son efficacité à des températures et des pressions physiologiques. À l'avenir, les chercheurs pourraient appliquer ces connaissances à la conception d'enzymes artificielles utilisées dans l'industrie chimique.
Note: Cet article a été traduit à l'aide d'un système informatique sans intervention humaine. LUMITOS propose ces traductions automatiques pour présenter un plus large éventail d'actualités. Comme cet article a été traduit avec traduction automatique, il est possible qu'il contienne des erreurs de vocabulaire, de syntaxe ou de grammaire. L'article original dans Anglais peut être trouvé ici.
Publication originale
Takeshi Murakawa, Kazuo Kurihara, Mitsuo Shoji, Naomine Yano, Katsuhiro Kusaka, Yoshiaki Kawano, Mamoru Suzuki, Yasuteru Shigeta, Takato Yano, Motoyasu Adachi, Katsuyuki Tanizawa, Toshihide Okajima; "Neutron Crystallography of a Semiquinone Radical Intermediate of Copper Amine Oxidase Reveals a Substrate-Assisted Conformational Change of the Peptidyl Quinone Cofactor"; ACS Catalysis, Volume 13, 2023-9-7